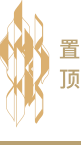生物信息学和人工智能平台
北京市感染性疾病研究中心建有生物信息学和人工智能技术平台。该平台在硬件、软件和人员方面提供了满足生物信息学分析和人工智能相关程序开发所需的支撑。具体包括:
1. 硬件平台方面
为满足生物信息学和人工智能技术开发需求,平台配备由高性能CPU计算平台、高性能GPU计算平台和高性能存储平台组成的硬件支撑平台。该平台可以满足临床和科研所需的基因组学、转录组学(单细胞)、蛋白质组学、代谢组学分析,以及大语言模型知识库构建、模型微调、智能体开发等需求。
(1)高性能CPU计算平台(用于生物信息学分析)
计算节点使用AMD EPYC 7763处理器(64核,主频2.45GHz)处理器,单节点不少于1024GB DDR4内存,支持GPU与CPU协同计算。提供不少于25TFLOPS的计算能力,可并行处理2000+线程的计算任务。
(2)高性能GPU计算平台(用于人工智能开发)
配置8块NVIDIA A100 Tensor Core GPU(80GB显存),支持NVLink互连技术,提供每秒312TFLOPS的混合精度计算能力,适用于深度学习模型训练(如卷积神经网络、Transformer)和千亿参数模型的分布式训练(如Megatron-LM、DeepSpeek框架)和本地化推理。
(3)高性能存储平台
采用企业级并行存储系统,总容量≥2PB,支持对象存储与块存储混合模式,满足多组学数据的长期归档与快速检索需求。配备全闪存阵列(All-Flash Array)作为热数据存储层,提供每秒200万IOPS和100GB/s吞吐量,适配实时分析场景,集成RAID冗余与异地灾备机制,结合访问审计系统,符合临床研究伦理要求
2. 软件平台方面
(1)生物信息学支撑平台
该平台集成了覆盖基因组学、转录组学、蛋白质组学和代谢组学全流程的开源与自主研发分析工具,不仅支持各单组学的深度解析,更能实现跨组学的系统整合与联合挖掘。在基因组学方面,部署了BWA、GATK等标准工具以及自主研发的TracePatho致病菌分析和溯源系统等流程,支持从序列变异到代谢通路的全景式生物医学研究。所有工具均针对高性能计算环境优化,确保从原始数据到生物学洞见的高效转化。
(2)人工智能大语言模型的本地部署
实现了人工智能大语言模型的本地化部署,不仅支持通用领域的预训练大模型,还针对生物医学领域进行了深度优化与定制开发。在基础模型方面,部署了LLaMA-3、GPT-NeoX等开源架构;在专业领域强化方面,整合了PubMed文献库、临床诊疗指南和组学数据库进行持续训练,实现从文献挖掘到实验假设生成的智能辅助,确保数据隐私安全的同时提供媲美云端服务的响应速度,为科研创新和临床决策提供强大的智能支持。
(3)全国临床传染病信息网络系统
通过整合第三方医检机构(金域医学)和地坛医院感染专科联盟及哨点医院构建了覆盖全国的临床传染病信息网络系统。该系统的数据可以覆盖全国31个省、314个城市、4518家医疗机构,覆盖200种病原体(包括亚型),年可用样本量超过200万例。该信息网络系统的数据实时更新,数据时效性为T+2天。
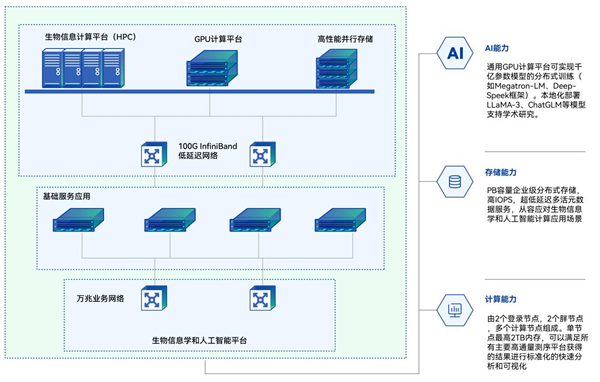
(4)常见致病菌分析和溯源系统
常见致病菌分析和溯源系统(TracePatho)是一款专为流行病学家、临床医生以及公共卫生研究人员设计的细菌全基因组分析平台,融合了27种关键医学病原体的基因组数据库。该平台支持血清型、MLST、cgMLST等多种分子分型方法,并结合菌株的详尽信息,以实现感染源的精确定位。TracePatho提供了一个简洁的操作界面和一系列可视化工具,有助于快速诊断和疫情研究的防控工作。(https://tracepatho.com/)
(5)呼吸道传染病防控专家机器人
利用DeepSeek作为大型模型的基础架构,通过检索增强生成技术(RAG)将29种传染病流行病学理论研究结果以知识库的形式赋予模型,利用超过300万例急性呼吸道传染病数据对模型进行训练,最终开发出了一款呼吸道传染病防控专家机器人。该机器人能够提供关于传染病疫情的实时问答服务,并进行疫情趋势的分析研判。
3. 平台开发及合作交流
北感中心的生物信息学和人工智能平台为开发平台,欢迎感兴趣的医院、高校和科研院所交流、合作和使用,有任何交流、合作和使用需求,可随时联系我们,联系方式为:nksunyamin@aliyun.con 18622172239
上一篇: 多组学平台








 京公网安备 11010502052111号
京公网安备 11010502052111号